Základy praktické bioinformatiky
Section outline
-
-
Toto jsou ukázkové domácí úkoly vypracované T. Gazárkovou.
-
-
-
-
-
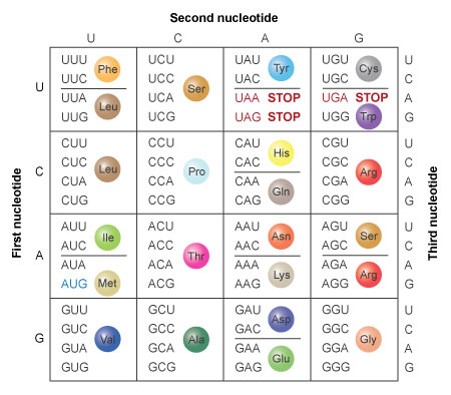
Návrh primerů
PCR primer design
.OligoCalc-pro kontrolu vlastností primeru (bez zadání sekvence)
Pro získání druhého řetězce použijte program v rámci SMS: Reverse Complement
Kontrola primerů: Multalin.
-
-
-
Zde budou zkouškové verze a prostor pro odevzdávání písemek.